Как боб, кот и трава-мурава следствию помогали
Первое преступление, раскрытое с помощью анализа нечеловеческой ДНК, произошло в 1993 году в Аризоне. Преступника обличило дерево, под которым нашли жертву. Его ствол был поврежден столкновением с грузовиком, а в кузове машины подозреваемого следователи нашли плоды такого же дерева, паркинсонии. Когда же они сравнили ДНК, то установили, что плоды принадлежат тому самому пострадавшему растению. Такое сопоставление индивидуальных генетических “узоров” — неважно, людей или деревьев — называется ДНК-фингерпринтингом.

Паркинсония цветущая отцвела и готова накидать вам бобов в кузов. Фото: sus_scrofa
Еще одно растение, которое выступало в суде в качестве свидетеля — птичий горец Polygonum aviculare, известный также как трава-мурава. Убийца увез тело жертвы подальше от дома и сбросил его в ручей. На берегу рос горец, и его семена прилипли к шинам автомобиля, где их и нашли криминалисты.
На первый взгляд, трава, в отличие от дерева, едва ли способна помочь следствию: не искать же на месте преступления ту самую травинку, чья ДНК обнаружилась на вещах подозреваемого. Однако горец размножается в основном вегетативно или с помощью самоопыления, поэтому обширные пространства заняты если не клонами, то очень близкими родственниками. На радость криминалистам, и перекрестное опыление между горцами тоже иногда случается — а оно создает новые комбинации ДНК и поэтому добавляет полянам уникальности. Так что сравнение ДНК показало, что семена принесены именно с места преступления. Чтобы исключить случайное совпадение, криминалисты собрали дополнительные образцы еще в десятке мест, куда ездил подозреваемый — но генетически близких горцов больше нигде не нашлось.
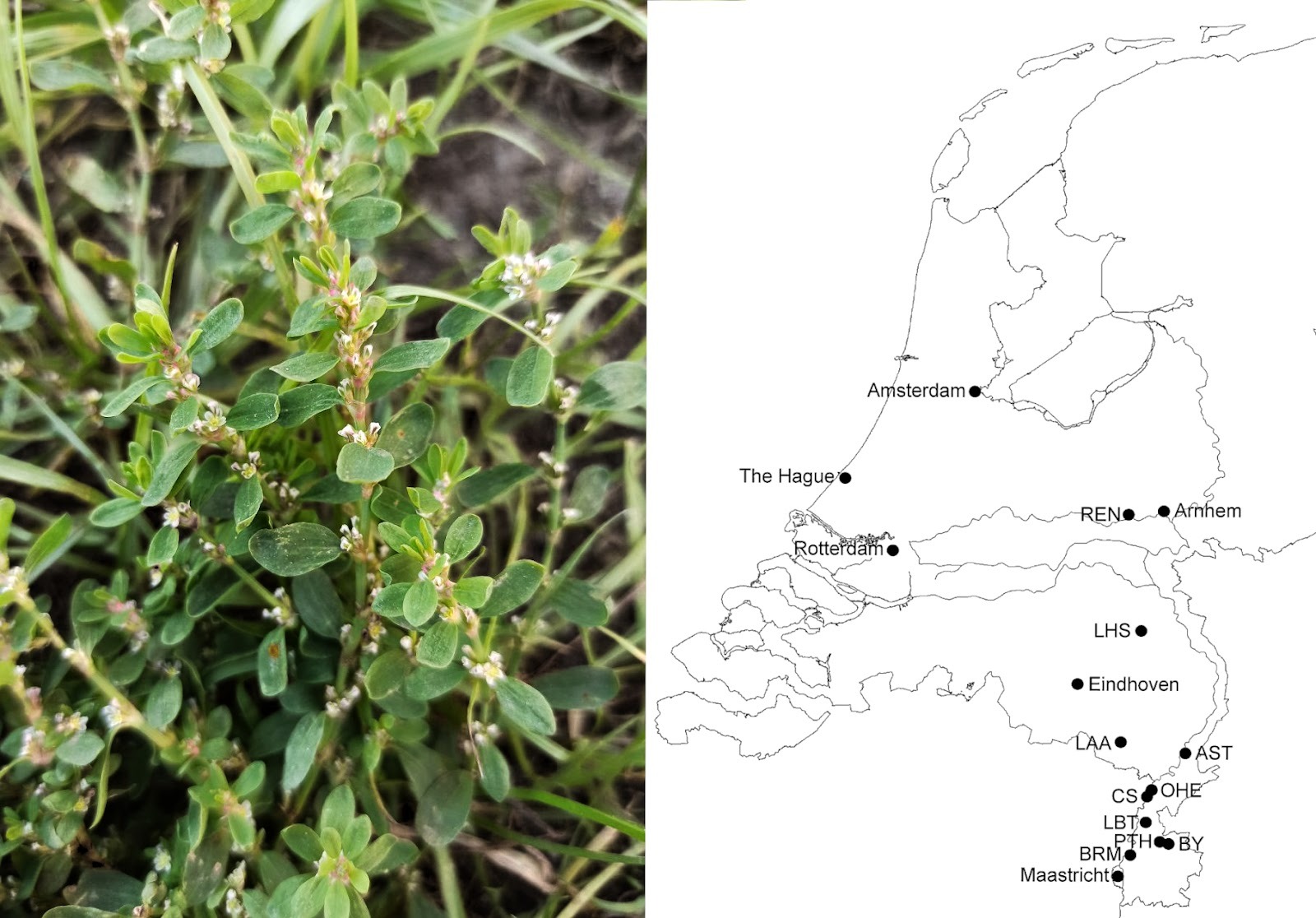
Горец птичий (фото автора) и карта, отражающая усилия голландских криминалистов по его секвенированию. CS — место преступления, BY — задний двор дома подозреваемого. Источник: [1]
Не только случайно встреченных растений следует опасаться преступнику: любимое домашнее животное тоже может его выдать. В 1994 году в восточной Канаде обнаружили тело убитой женщины. Главным подозреваемым был муж, вот только с уликами поначалу не заладилось. Но через несколько недель неподалеку обнаружили куртку, покрытую кровью жертвы, а на ней — волоски белой шерсти. Белой как Снежок — кот, принадлежавший мужу убитой.
Лабораторию, которая взялась бы сравнить ДНК кота с образцом, выделенным из шерстинок, следователи нашли не сразу; в итоге им помогли исследователи, занимавшиеся наследственными заболеваниями. Совпадение оказалось идеальным. Мужчина получил пожизненное заключение, а фингерпринтинг нечеловеческой ДНК — признание. С тех пор анализ геномов кошек и собак много раз помогал в расследованиях, а в некоторых странах для нужд криминалистов даже появились базы данных ДНК домашних животных.

Кошки отлично подходят на роль свидетелей обвинения. Обычно из шерстинки животного, лишённой волосяной луковицы, очень сложно извлечь ядерную ДНК; добыть из отмерших клеток хотя бы митохондриальную ДНК, более устойчивую, но менее информативную, — уже удача. Но именно у кошек есть полезная привычка: они постоянно вылизываются, а в слюне содержится множество живых клеток. Фото: Наталья Дьячкова, Дарья Иванова
Где искать идентификационный номер
В среднем у двух особей одного вида совпадает порядка 98-99 % генома. У двух случайно выбранных людей, например, различия составляют всего 0,4% — но это целых 5,5 млн генетических вариаций. Проблема в том, что они разбросаны по всему геному. В материале про пыльцу и криминалистику мы писали, что определить вид дерева можно всего по одному фрагменту длиной несколько сотен нуклеотидов — “штрихкоду” вида. А вот для опознания конкретного дерева приходится сопоставлять множество отдельных изменчивых участков ДНК (локусов).
В одном-единственном локусе варианты у двух деревьев вполне могут совпасть случайно. Но если проверить несколько независимых участков, то вероятность, что они случайно совпадут все разом, крайне мала — а точнее, равна произведению вероятностей одиночных совпадений. Для человеческого ДНК-фингерпринта из 13 локусов, который раньше был стандартом в США, эта вероятность меньше 10-14 [2]: едва ли у вас найдется хоть один неродной тринадцатилокусный “близнец” среди всего населения планеты.
Независимость локусов очень важна. Если они расположены в геноме слишком близко друг к другу, то будут часто наследоваться вместе, и 13 таких локусов дадут не намного больше информации, чем один. А вот более далёкие локусы “отцепляются” друг от друга во время полового размножения за счёт рекомбинации — перемешивания родительской ДНК. Понятно, что это не сработает, если родитель номер один совпадает с родителем номер два — именно поэтому хороший свидетель не должен ограничиваться самоопылением.
Если «штрихкод» вида у всех растений находится в одном месте, то универсальной карты изменчивых локусов, увы, не существует. Если вы первым захотели узнавать «в лицо» огурцы, то вам и придется искать в их геномах нужные локусы и подсчитывать, как часто встречается каждый из вариантов. Это понадобится, чтобы продемонстрировать в суде (не)вероятность ошибочного совпадения.
Чаще всего для идентификации — неважно, людей, котов или трав — используют STR, short tandem repeats. Как следует из названия, эти локусы состоят из множества коротких, от одного до шести нуклеотидов, повторов. Мутации в них происходят на несколько порядков чаще, чем в среднем, потому что повторы сбивают с толку копирующий фермент (ДНК-полимеразу): она может случайно проскользнуть на несколько повторов вперед или назад и продолжить работу с неправильного места. В результате локус либо укорачивается, либо прирастает дополнительными повторами, так что в популяции может быть распространено до 20 версий с разным числом повторов. А чем больше изменчивость, тем меньше локусов нужно для идентификации! Несмотря на то, что STR могут составлять до 3 % всего генома, они обычно не функциональны: не кодируют белки и ничего не регулируют, а потому мутации в них не влияют на организм и невидимы для естественного отбора. Эта нейтральность — тоже важное свойство для дискриминирующего локуса, потому что отбор меняет частоты вариантов (хорошие распространяются, плохие — исчезают), и расчеты теряют надёжность.
Как STR стали стандартом
STR удобны еще и тем, что их не обязательно секвенировать, чтобы определить вариант – достаточно измерить длину. Первый подходящий для этого метод, Саузерн-блоттинг, появился еще до изобретения секвенирования. Работает он так: ДНК нарезают ферментами-”ножницами” (рестриктазами), которые узнают конкретные короткие последовательности (скажем, АТТА). Затем эту нарезку разделяют с помощью электрофореза: попав в электрическое поле, отрицательно заряженная ДНК ползет сквозь гель к положительно заряженному полюсу, и чем короче фрагмент, тем дальше он успевает уползти за отведенное время. Чтобы увидеть среди всей массы фрагментов те, что содержат нужные локусы, их раньше метили комплементарным радиоактивным зондом.
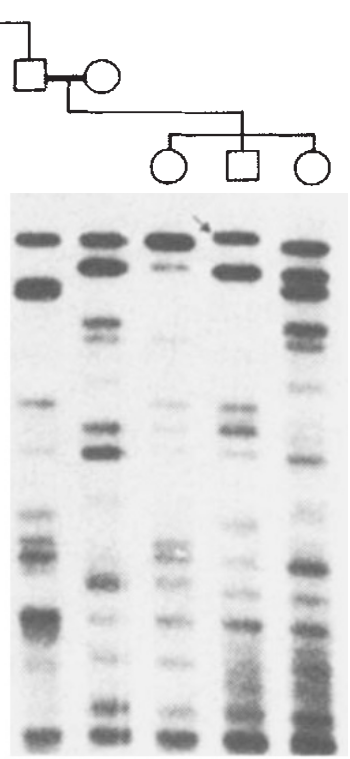
Родители и дети, Jeffreys et al.,1985. Фингерпринт на рентгеновской пленке. Каждая ступенька “лесенки” унаследована детьми (справа) либо от матери, либо от отца (слева). Хотя постойте, не каждая: стрелочкой помечен новый мутантный вариант. Он чуть выше, чем любой из родительских — значит, в нем больше повторов. Не пытайтесь разобраться, что происходит в нижней части: разрешения гель-электрофореза не хватает, чтобы получить четкую картинку для коротких фрагментов. Источник [3]
Сейчас вместо разрезания ДНК используют размножение нужных фрагментов (ПЦР) — это позволяет работать с минимальными количествами биоматериала. А теплую радиоактивную технологию вытеснили флуоресцентные зонды. Причем свечение автоматически регистрируется прибором, и исследователь видит на экране только холодные цифровые пики:
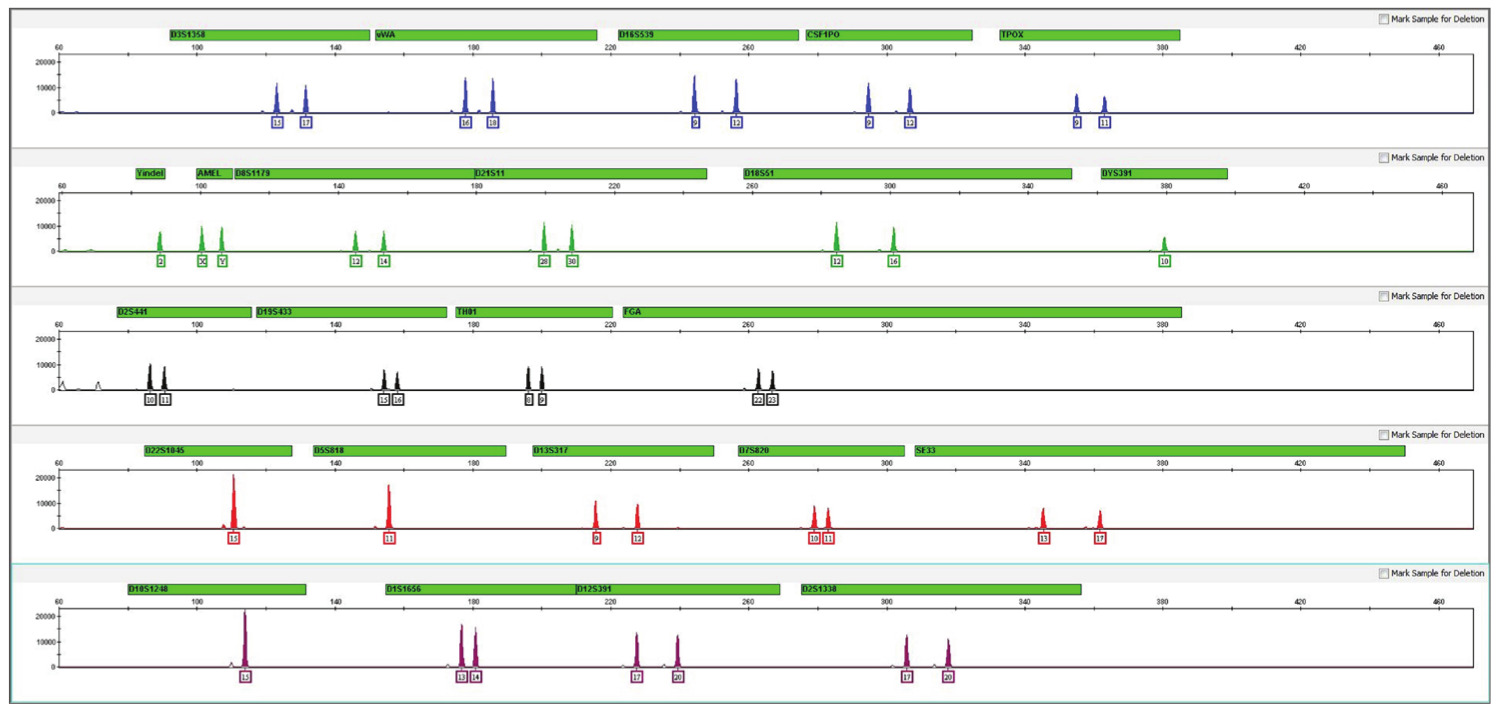
Фингерпринт для 24 локусов: по вертикали — интенсивность свечения, по горизонтали — длина фрагмента. Если в блоке один высокий пик — организм унаследовал одинаковые варианты локуса от обоих родителей, если два пика рядом — отцовский и материнский варианты различаются по длине (источник). Это фингерпринт простого человеческого диплоидного генома, но многие растения — полиплоиды, то есть у них не два набора хромосом, а, скажем, 4 или 6. Соответственно, и разных вариантов локуса у одного растения может быть больше двух.
A сложные для чтения, но красивые флуоресцентные “лесенки” до сих пор появляются в кино как иллюстрация ДНК-фингерпринтинга:
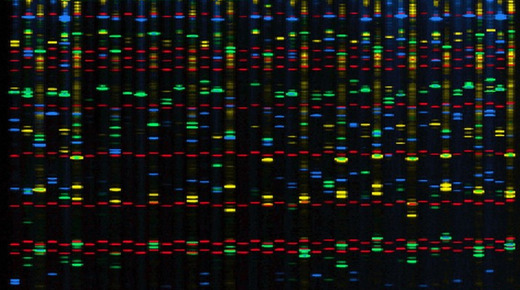
Зонды для удобства красят в разные цвета. Красным светятся линейки — маркеры известной длины (источник).
Впрочем, есть у STR-локусов и недостатки: они бывают длинными, а длинные последовательности быстрее разрушаются. Их сложнее поймать, если ДНК плохо сохранилась или ее совсем мало, что актуально при работе с древесиной — ведь это мертвая ткань. Да и высокая частота мутаций — не всегда благо (например, в тестах на отцовство новые мутации только мешают). К счастью, технологии секвенирования так прокачались, что прочесть последовательность локуса — притом не одного, а сразу многих — стало не намного сложнее, чем определить длину. Так что сейчас к стандартным наборам STR-локусов могут добавлять SNP-локусы (single nucleotide polymorphisms), то есть обычные замены одной буквы на другую. Пусть они менее изменчивы, зато подходят для сложных случаев.
Как увидеть за деревьями лес: геолокация по ДНК
Анализ ДНК помогает и в тех случаях, когда жертвой оказались сами растения или животные. Оставленные на вырубке пни могут стать решающими в расследовании браконьерства: если найдутся срубленные деревья, их можно сопоставить с пнями по ДНК. Так удалось доказать вину нарушителей, уничтоживших клены в национальном парке Олимпик (Вашингтон, США), и защитить от нелегальной рубки кипарисовики на Тайване. Иногда идентифицировать дерево можно и без ДНК: например, если удалось перехватить необработанные бревна, можно сличить спилы; но ДНК-фингерпринтинг позволяет опознать даже доски и опилки.
Даже если не найдено ни рожек, ни ножек, ни пней, ДНК все равно может дать подсказку, указав на вероятный район происхождения добычи. Браконьерство часто годами остается незамеченным, особенно в диких и сложных для наблюдения, но уязвимых местах — таких, например, как дождевые леса. Выяснив, откуда привезена древесина или слоновая кость, можно вовремя заметить популяции, которым угрожает опасность. А еще методику берут на вооружение археологи. Если понять, где выросло дерево, пошедшее на строительство старых зданий или затонувших кораблей, это прояснит историческое расположение лесов и торговых путей.
Идея та же, что в “человеческих” генетических тестах на происхождение, таких как тесты от Genotek или 23andMe. Только коммерческие тесты решают задачу классификации, то есть пытаются соотнести вас с референсными популяциями (“вы на 5 % похожи на ашкенази, и на 20 % – на скандинавов”), а для криминалистов полезнее может оказаться другой подход – регрессия, то есть прямое предсказание географических координат по ДНК. Деревья — более удобный объект для такого исследования, чем люди: они перемещаются намного медленнее (хотя люди и тут всё могут запутать, потому что уносят некоторые яблочки на другой континент от яблони).
Для геолокации в идеале нужны уже не десятки локусов, а хотя бы сотни — чем больше, тем лучше. Предсказывать координаты можно множеством методов: от простейшего kNN до нейросетей (LOCATOR) и регрессии гауссовского процесса. В последнем подходе в явном виде зашита идея, что у похожих генотипов координаты коррелируют сильнее, а еще он сразу определяет надежность предсказания. Есть и геолокационные методы, основанные на моделях популяционной генетики (SPASIBA), но они, как ни странно, справляются хуже более общих; к тому же они способны работать только с генетическими данными. А вот какой-нибудь случайный лес (здесь читателю предлагается поиграть со словами самостоятельно) способен вдобавок к ДНК учесть соотношение ширины годовых колец, химический и изотопный составы древесины, зубов, чешуи или шерсти. Кстати, эти данные указывают именно на регион обитания, а не на регион происхождения – а для искусственно высаженных растений или заново интродуцированных животных эти регионы могут различаться.
Обучающие выборки пока малы и неравномерны, так что и результаты не слишком впечатляют: например, в недавнем исследовании медианная ошибка при геолокации бука европейского составила 55 км – это примерно 2% от протяженности его европейского ареала. Но и этого может оказаться достаточно, чтобы подтвердить нелегальный импорт древесины или направить рейды против браконьерства в нужный регион [4].
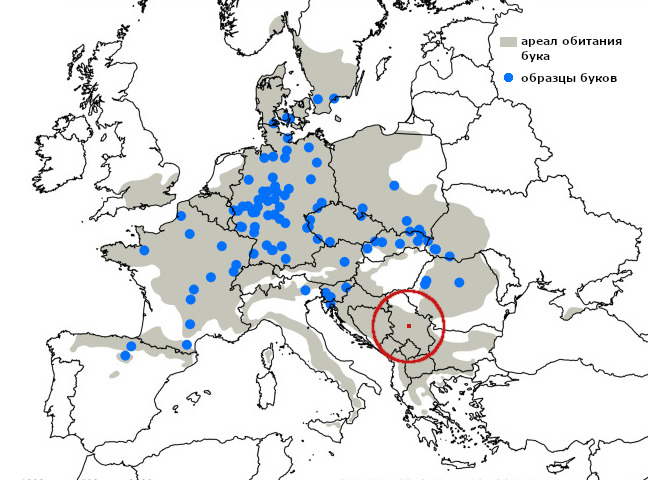
Синие точки — обучающая выборка, 865 деревьев с известными координатами и данными для 30 000 SNP-локусов. Красная окружность показывает масштаб ошибки: для 90% буков расстояние между предсказанной и реальной локацией — меньше 8% от протяженности ареала, то есть около 250 километров. Правда, для балканских буков ошибка должна быть еще больше, потому что их вовсе нет в выборке (источник).
Геолокация по ДНК едва ли когда-нибудь станет точной: даже если совпадение почти полное, нельзя гарантировать, что дуб не привезли желудем с другого конца света. Но можно секвенировать столько дубов, что задача вплотную приблизится к сверке образца с базой. На создание таких обширных генетических баз данных — не только для людей, но и для домашних животных, и для ценных пород деревьев — и делают ставку криминалисты. Похожую базу можно было бы создать и для потенциальных свидетелей — например, малозаметных растений-космополитов, таких как горец.
Индивидуальные ДНК-паттерны растений помогают искать не только убийц и браконьеров. Например, с их помощью также можно проверить сорт оливок, из которого произведено масло, или определить источник наркотиков растительного происхождения. В перспективе, если человечество не задумается о защите персональных данных растений, каждая доска и каждый в поле колосок будут иметь генетический паспорт с указанием происхождения, который невозможно подделать.