Чтобы остановить эпидемию, нужно вовремя ее заметить и понять, как она развивается. Для этого эпидемиологам нужны точные и свежие данные о заболеваемости. Как их собрать? Мы уже писали о том, как в 1854 году во время вспышки холеры в Лондоне врач Джон Сноу обошел дома в округе, нанес случаи заболевания на карту и выявил источник инфекции. Большинство данных и сегодня поступает от системы здравоохранения, пусть и более централизованно — через клиники и больницы. Но с появлением социальных сетей эпидемиологи получили еще один источник информации: теперь можно анализировать, что люди рассказывают о своем здоровье в интернете.
Публикации в X, посты в Facebook*, комментарии на форумах, поисковые запросы, отзывы на товары — все это позволяет в режиме реального времени отслеживать симптомы заболеваний и даже предсказывать вспышки.
Где искать эпидемиологические данные
Twitter (сейчас X) успел вдохновить множество эпидемиологических исследований благодаря огромному потоку постов и относительно открытой политике доступа к данным (в 2023 году она, увы, изменилась). Формат твитов идеально подходит для выявления ключевых тем (topic detection), а геотеги позволяют привязывать сообщения о симптомах к конкретным регионам и замечать необычные скопления жалоб. Более того, по геотегам можно изучать перемещения пользователей, что помогает моделировать распространение инфекции.
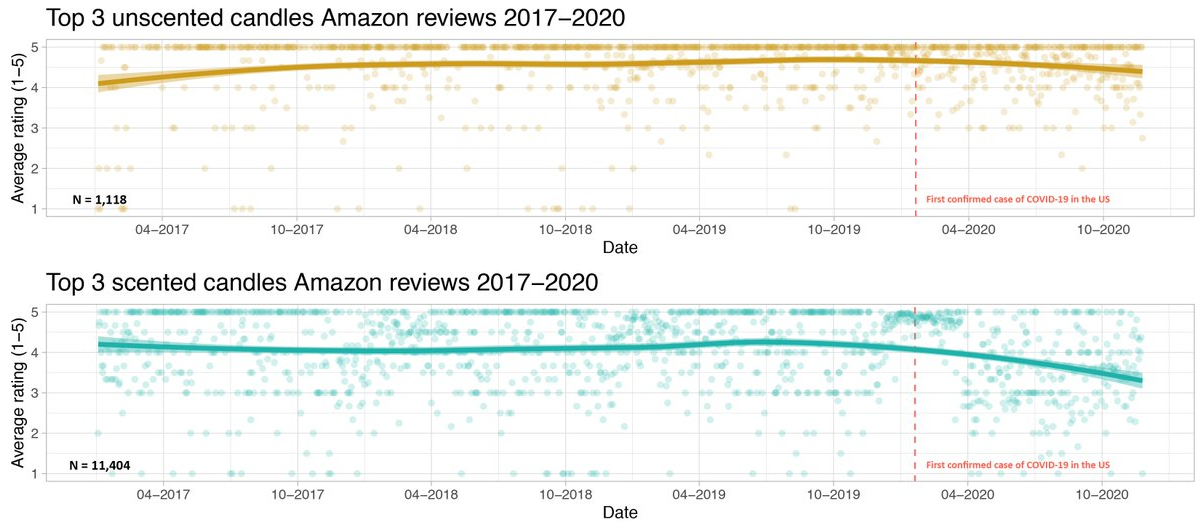
Но эпидемиологов интересуют и любые другие популярные открытые платформы, где люди оставляют сообщения. Даже отзывы на товары иногда содержат информацию о симптомах — причем заболевший сам может не подозревать о своей болезни. Например, в начале пандемии COVID-19 покупатели стали оставлять больше негативных отзывов об ароматизированных свечах, возмущаясь, что они не пахнут.

Динамика поисковых запросов тоже позволяет увидеть, как растет интерес к определенным болезням в различных регионах. Из-за отсутствия контекста запросы могут быть более «шумными», чем твиты, однако информации в них больше: все-таки люди гораздо чаще (и раньше) гуглят симптомы, чем пишут о них посты.
Наконец, социальные сети вроде Facebook* или ВКонтакте содержат информацию о структуре контактов в обществе. Зная, как устроена карта связей между людьми, эпидемиологи могут точнее предсказать, как быстро будет распространяться инфекция.
Как найти в сети ранние признаки эпидемии
Идея проста: если в каком-то регионе люди массово начинают искать информацию о кашле, температуре и боли в горле или писать о симптомах в соцсетях, это может быть самым ранним сигналом о начале вспышки конкретного заболевания. В отличие от официальной статистики, которая всегда немного запаздывает, такую оценку можно получать в режиме реального времени.
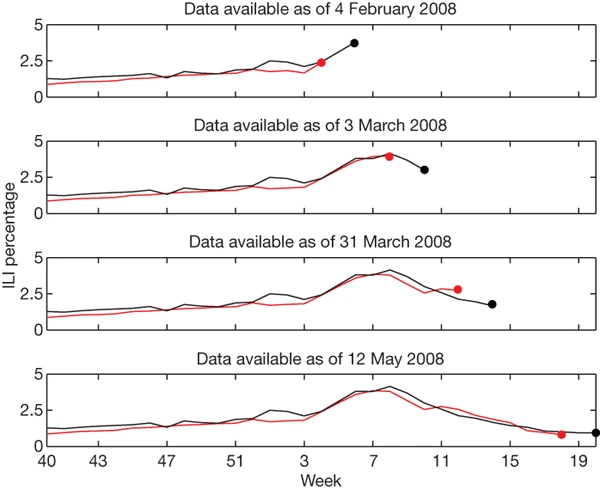
Еще в 2009 году было показано, что число поисковых запросов в Google может отражать число людей, болеющих гриппом. Исследователи выделили несколько десятков запросов, которые поодиночке лучше всего коррелировали с официальным числом заболевших, — например, «лечение простуды» и «осложнения при гриппе». Их комбинация позволила создать модель, которая оценивала динамику распространения вируса быстрее, чем Центр по контролю и профилактике заболеваний [1].
Для SARS-CoV-2 методика тоже работает. Например, в 2020 году в разных провинциях Китая число «ковидных» поисковых запросов в Baidu и постов в Weibo хорошо коррелировало с ежедневным приростом подтвержденных случаев. По данным разных исследований, пик запросов везде случался примерно за 3–20 дней до пика официально зарегистрированных заболеваний. Интересно, что для запроса «температура» опережения не было. Вероятно, это связано с китайской политикой повсеместной проверки температуры и немедленной изоляции людей с лихорадкой [2, 3].
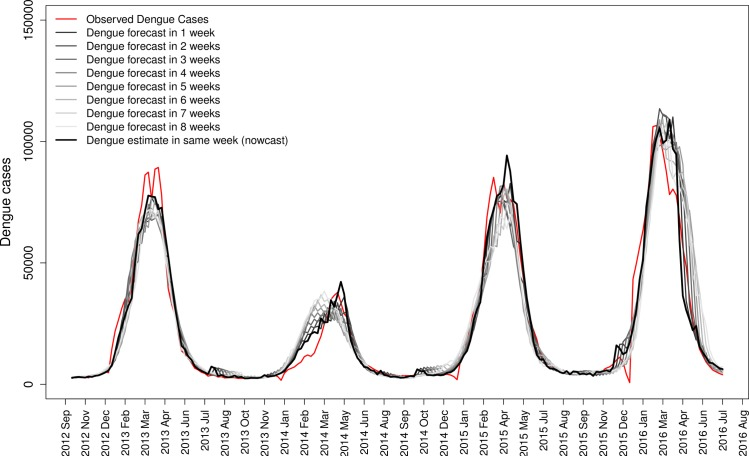
Подобные разработки особенно ценны для бедных регионов вроде бразильских фавел. Несмотря на проблемы с инфраструктурой, интернет и смартфоны там широко распространены, и люди, заболев, скорее выложат пост о плохом самочувствии, чем пойдут к врачу. Из-за этого инфекции часто распространяются незаметно для служб здравоохранения, а официальная статистика о количестве заболевших отстает на 3–4 недели. Бразильские ученые создали систему, которая на основе данных Twitter могла регистрировать зарождающиеся вспышки лихорадки денге. Она находила твиты по ключевым словам, отсеивала нерелевантные — например, шутки или обсуждения новостей — и по их числу оценивала число болеющих в реальном времени (nowcasting), а также предсказывала динамику на ближайшие недели. Так как денге — сезонное заболевание, модель дополнительно учитывала время года. Наблюдать за ходом эпидемии можно было не только на уровне страны, но даже на уровне городов с населением менее 100 тысяч человек [4].
Поисковые запросы помогают выявлять и совсем локальные вспышки — и даже определять их источник. Оказывается, с помощью агрегированных анонимных геоданных можно связать недавнее посещение конкретного кафе или ресторана с запросами, намекающими на отравление [5]. Когда санитарные службы шли по таким наводкам, нарушения обнаруживались в три раза чаще, чем в заведениях, выявленных стандартными методами. Даже прямые жалобы от клиентов работали хуже. Вероятно, дело в том, что примерно в трети случаев проблема была не в последнем посещенном месте, на которое обычно и жалуются, а в одном из предыдущих.
Связи решают все
«Системный Блокъ» уже писал о том, как анализ социальных сетей помогает выяснить, кого из недавних контактов заболевшего нужно тестировать в первую очередь. Но и течение эпидемии в целом напрямую зависит от того, как люди взаимодействуют друг с другом. Поэтому модели, которые учитывают структуру социальных связей, должны давать более точные предсказания.
Например, две важные характеристики сети связей — это величина плотно связанных подгрупп (то есть таких, где почти все знакомы друг с другом) и то, насколько хорошо эти подгруппы связаны между собой. Внутри каждой из них болезнь может распространяться быстро, а между подгруппами перекидывается реже. Поэтому размеры подгрупп и степень связности между ними определяют динамику эпидемии: распространение вируса в сети с кластерной структурой может происходить медленнее и достигать меньшего пика, чем при равномерном перемешивании. Наличие в сети «хабов» — людей с большим числом связей — тоже меняет динамику. По сравнению с сетью, где число контактов распределено более равномерно, общее число заболевших в итоге может оказаться меньше, зато пик эпидемии — выше. На ранних этапах «хабы» быстро заражаются и ускоряют распространение вируса. Однако после того как они приобретут иммунитет, эпидемия может сравнительно быстро закончиться [6].
По геотегам можно восстановить еще одну сеть — сеть связей между регионами. «Толщина» связей определяется, например, количеством дружеских контактов между пользователями из двух городов (SCI — social connectedness index), или числом пользователей, которые за определенный период отметились в обоих городах (PCI — place connectedness index). По этим данным можно предположить, как заболевание будет распространяться от одного региона к другому. Например, географическое распространение COVID-19 неплохо коррелировало с характеристиками сети, измеренными на основе Facebook* [7, 8] и Twitter [8].
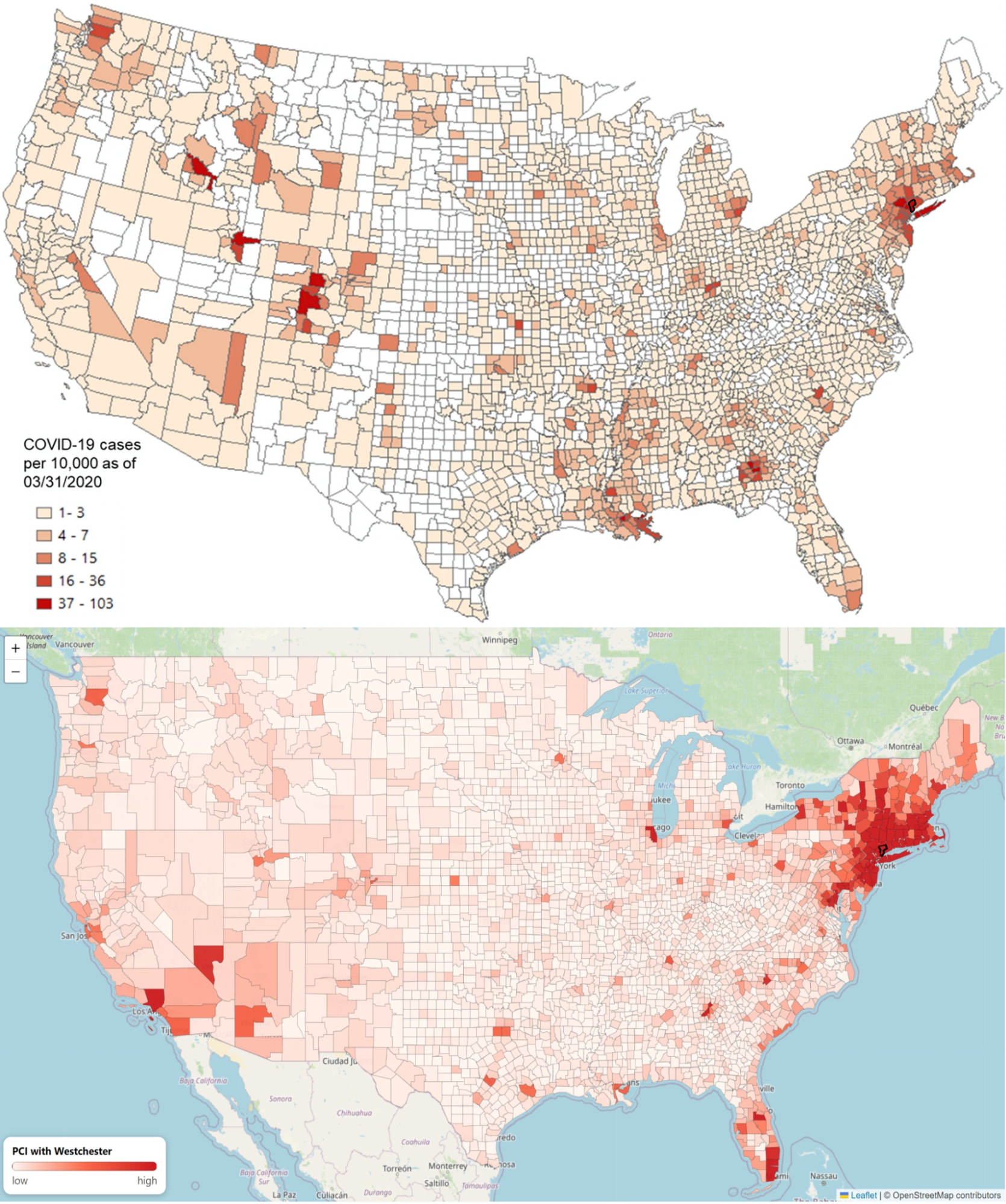
Проблемы и сложности
Модель Google Flu Trends, которая работала с поисковыми запросами о простуде, не смогла вовремя заметить пандемию гриппа H1N1 в 2009 году. Модель обновили, но это не помешало ей сильно завысить пиковую заболеваемость гриппом в сезоне 2012–2013, после чего проект был закрыт [9]. В чем же была проблема?
Поведение пользователей быстро меняется, поэтому простые модели не могут работать стабильно без постоянной перекалибровки. Например, когда всем стало известно о потере обоняния при COVID-19, стали появляться отзывы, которые сложно корректно обработать поиском по ключевым словам: «Свеча совсем не пахнет — и нет, у меня нет ковида». Шум тоже изменчив. Сезонный всплеск упоминаний «предвыборной лихорадки» или вирусный трек группы «Приступ бронхита» — и красивой корреляции конец. С такими проблемами помогает тщательный анализ контекста с помощью NLP — которого в Google Flu Trends не было. Но есть и более фундаментальное ограничение: медийность самого заболевания сильно влияет на то, как часто его симптомы будут искать и обсуждать, и от таких искажений NLP не спасет.
Вдобавок к этому, пользователи социальных сетей — не самый надежный источник сведений. Даже если они жалуются именно на грипп, едва ли они сдавали тест, чтобы отличить его от более распространенных рино- и коронавирусов. Кроме того, существует демографическое искажение: разные возрастные и социальные группы представлены в соцсетях неравномерно.
Наконец, подвижен сам цифровой ландшафт. Платформы рождаются, набирают популярность, меняют API и умирают, а пользователи начинают обсуждать симптомы с LLM, вместо того чтобы гуглить их. Поэтому любой проект цифрового эпидемиологического надзора должен непрерывно поддерживаться и обновляться.
Объединяй и здравствуй
Две основные тенденции последних лет — объединение разнородных цифровых следов и применение ИИ для их анализа. Пандемия COVID-19 породила волну исследований, в которых используются не только уже упомянутые источники, но и travel-блоги, трафик Википедии, данные новостных сайтов, информация от носимых медицинских устройств и поисковые запросы врачей (например, такую статистику собирает предназначенный для специалистов сайт UpToDate). Многие модели учитывают и более привычные для эпидемиологов данные: мобильность, сезонность, климатические и социодемографические факторы, наконец, официальные отчеты о заболеваемости за предыдущие дни [2, 10].
Интеграция множества источников делает модель устойчивее к переобучению. Например, в одном из исследований для обнаружения новых вспышек COVID-19 прокси-данные за последние две недели — скажем, число гугл-запросов «кашель» — аппроксимировались экспоненциальной кривой. Значимо восходящая кривая считалась признаком вспышки. Объединенные показания всех типов данных (гармоническое среднее разных p-value) имели лучшую чувствительность и точность, чем любой из них по отдельности. В качестве одного из прокси были взяты предсказания популярной эпидемиологической модели GLEAM. Она моделирует развитие эпидемии, учитывая пространственную структуру популяции, активность перемещений между разными локациями и устройство сети контактов для разных возрастных групп. Любопытно, что предсказания GLEAM оказались наименее чувствительным предиктором зародившейся вспышки, а наилучшими были поисковые запросы медиков [10].
Предсказывать динамику по нескольким источникам тоже пробовали. Например, исследователи из Германии адаптировали описанную выше схему для немецкого языка, объединили данные Google и Twitter и обнаружили, что с помощью LSTM-модели тренд заболеваемости можно прогнозировать на две недели вперед [11].
Публикацией исследований дело не ограничивается: появились платформы, которые автоматически анализируют релевантные источники с помощью NLP и постоянно ищут признаки новых вспышек. Среди них — проект ВОЗ EIOS (The Epidemic Intelligence from Open Sources), который создан для помощи службам здравоохранения, и коммерческая платформа EPIWATCH. А HealthMap отображает собранную в интернете информацию о заболеваниях, в том числе неинфекционных, на публично доступной карте (похожую карту, с выдержками из медиа, можно увидеть и у EPIWATCH).
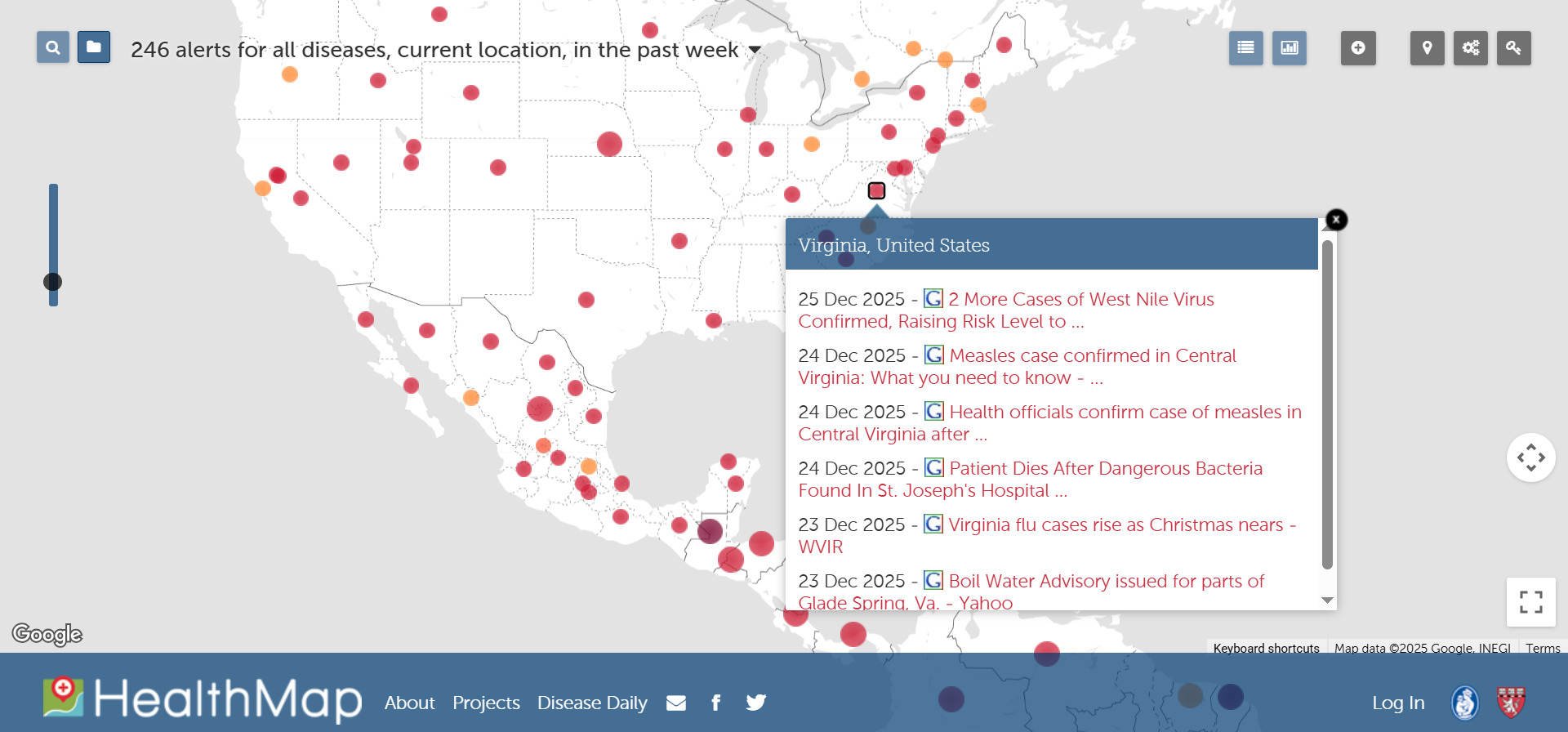
Оценить работу таких систем можно по недавнему отчету о ранней детекции заболеваемости в африканских странах: EIOS зарегистрировал 81% вспышек, информация о которых официально дошла до ВОЗ, из них 47,4% — до официального оповещения. В 2018 году, когда система только появилась, таких ранних обнаружений было меньше, 44,1% (кстати, зарождающуюся пандемию COVID-19 система вовремя не отследила) [12].
Будущее интернет-эпидемиологии
Интернет-эпидемиология — динамичное направление, которому еще предстоит адаптироваться и к наступившей ИИ-реальности, и, возможно, к увеличению числа носимых медицинских устройств. Если вчера, чтобы эпидемиологи услышали ваш голос, приходилось писать в Твиттер, то, может быть, уже завтра достаточно будет характерным образом закашлять рядом с умной колонкой, чтобы эпидемиологи за вами выехали. Это станет шагом к более безопасному миру!
*принадлежат компании «Meta», признанной экстремистской организацией и запрещенной в РФ
Источники
1. Ginsberg J., Mohebbi M.H., Patel R.S. et al. Detecting influenza epidemics using search engine query data // Nature. 2009. Vol. 457. P. 1012–1014.
2. McClymont H., Lambert S.B., Barr I. et al. Internet-based surveillance systems and infectious diseases prediction: An updated review of the last 10 years and lessons from the COVID-19 pandemic // J. Epidemiol. Glob. Health. 2024. Vol. 14. P. 645–657.
3. Tu B., Wei L., Jia Y., Qian J. Using Baidu search values to monitor and predict the confirmed cases of COVID-19 in China: — evidence from Baidu index // BMC Infect Dis. 2021. Vol. 21. Article number: 98.
4. Marques-Toledo C. de A., Degener C.M., Vinhal L. et al. Dengue prediction by the web: Tweets are a useful tool for estimating and forecasting Dengue at country and city level // PLoS Negl. Trop. Dis. 2017. Vol. 11. P. e0005729.
5. Sadilek A., Caty S., DiPrete L. et al. Machine-learned epidemiology: real-time detection of foodborne illness at scale // NPJ Digit. Med. 2018. Vol. 1. Article number: 36.
6. Pérez-Ortiz M., Manescu P., Caccioli F. et al. Network topological determinants of pathogen spread // Sci. Rep. 2022. Vol. 12. Article number: 7692.
7. Kuchler T., Russel D., Stroebel J. JUE Insight: The geographic spread of COVID-19 correlates with the structure of social networks as measured by Facebook // J. Urban Econ. 2022. Vol. 127. P. 103314.
8. Li Z., Huang X., Ye X. et al. Measuring global multi-scale place connectivity using geotagged social media data // Sci Rep. 2021. Vol. 11. Article number: 14694.
9. Aiello A.E., Renson A., Zivich P.N. Social media- and Internet-based disease surveillance for public health // Annu. Rev. Public Health. 2020. Vol. 41. P. 101–118.
10. Kogan N.E., Clemente L., Liautaud P. et al. An early warning approach to monitor COVID-19 activity with multiple digital traces in near real time // Sci. Adv. 2021. Vol. 7. N. 10. DOI: 10.1126/sciadv.abd6989.
11. Wang D., Lentzen M., Botz J. et al. Development of an early alert model for pandemic situations in Germany // Sci. Rep. 2023. Vol. 13. Article number: 20780.
12. Williams G.S., Koua E.L., Abdelmalik P. et al. Evaluation of the epidemic intelligence from open sources (EIOS) system for the early detection of outbreaks and health emergencies in the African region // BMC Public Health. 2025. Vol. 25. Article number: 857.