В 1969 году под Веной во время сплава по Дунаю пропал человек. Следователи арестовали подозреваемого, у которого был мотив для убийства, но не могли добыть доказательств его вины — пока не отправили образец грязи с его ботинок специалисту по пыльце (палинологу). Тот нашёл в образце пыльцу ивы, ели, ольхи, а также давно вымершего в Европе дерева гикори. По словам эксперта, такой букет — так называемый пыльцевой спектр — можно было собрать только в одном месте Австрии: в небольшом регионе в 20 километрах к северу от Вены, где обнажались отложения миоценового периода — оттуда и вымыло окаменевшую пыльцу гикори, — и где рос елово-ольхово-ивовый лес. Узнав, что криминалисты могут скоро раскрыть преступление, подозреваемый сознался и показал место, где было спрятано тело [1].
Почему пыльца удобна для геолокации
Пыльца незаметна и крайне устойчива. Ни 20 миллионов лет, ни стирка с отбеливателем не избавят вашу одежду от её следов полностью: спорополленин, покрывающий пыльцевое зерно, — одна из самых химически стабильных органических субстанций, а из-за малых размеров (в среднем 20–60 микрометров) и выростов на поверхности пыльца надёжно запутывается в волокнах ткани.
Пыльцевые зёрна ещё и уникальны. По их внешнему виду можно определить, каким растениям они принадлежат — значит, можно выяснить и регион происхождения образца. Это помогает отследить трафик наркотиков и ввоз санкционных товаров или доказать, что бабочка-репейница, которую вы встретили на побережье Испании, зимовала в Африке и побывала в Сахаре [2]. Даже в пределах одной природной зоны растительные сообщества сильно различаются: в лесу, городском парке и в пойме реки пыльцевые спектры будут разными.
Но определить видовую принадлежность растения, глядя на пыльцу, не так просто. Раньше это мог сделать только специалист-палинолог. Сейчас появились системы автоматической классификации по микрофотографиям (например, PIGLT [3]), и уже не обязательно искать эксперта и пересылать ему образцы в конверте; но фотографии делать всё же придется, а это тоже дело долгое и трудоёмкое. Есть и другая проблема: часто по внешним признакам пыльцы в принципе невозможно определить растение с точностью до вида. А хотелось бы! Ведь чем точнее классификация, тем более узок регион поиска.
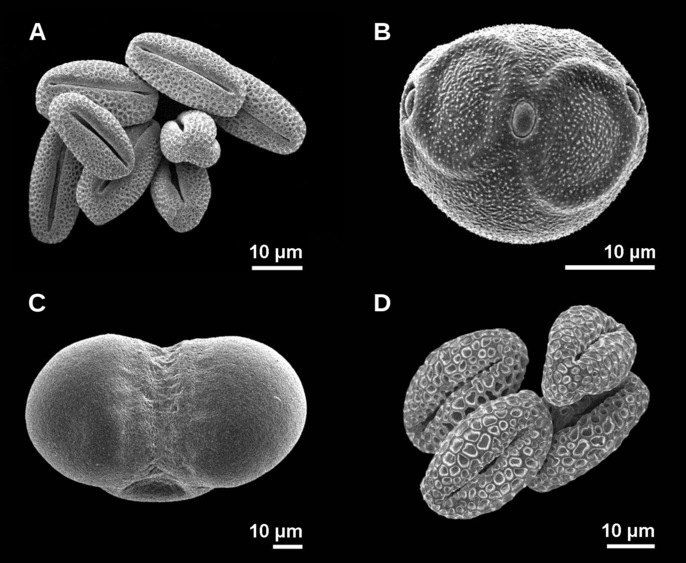
Рисунок 1. Пыльца ивы козьей (Salix caprea, A), ольхи чёрной (Alnus glutinosa, B), ели обыкновенной (Picea abies, C) и сирени обыкновенной (Syringa vulgaris, D) [4–7]
Как определить вид растения (и не только) с помощью ДНК
К счастью, теперь видовую принадлежность можно определить с помощью секвенирования ДНК. Больше того, не пыльцой единой покрыты вещи: обломки сухих листьев, одинаковые на вид гифы грибов, бактерии, — человеку, вооружённому секвенатором, каждая букашка может помочь в поиске. Чтобы определить вид образца, прицельно прочитывают небольшой максимально информативный фрагмент его генома, так называемый баркод, а затем сравнивают его с известными баркодами из базы данных.
Фрагменты, способные сыграть роль баркодов, исследователи выбирали не один год. Подходящих кандидатов не так много: одной стороны, такой участок должен присутствовать у всех организмов выбранной таксономической группы (например, у всех растений), то есть быть достаточно важным, чтобы от него нельзя было избавиться в ходе эволюции, а с другой — он должен различаться даже у близкородственных видов. Для этого ему нужно заниматься тем, чего важные участки обычно избегают: активно накапливать мутации. Кроме того, участок должен легко копироваться методом ПЦР (полимеразной цепной реакции), а для этого окружающие его последовательности, наоборот, должны быть очень похожи у всех организмов группы. Сам же он должен быть коротким, не более 1000 нуклеотидов.
На самом деле требований больше, и одного фрагмента не всегда достаточно для точного определения вида, поэтому в качестве баркода могут использовать последовательности двух или трёх фрагментов. У бактерий и архей обычно секвенируют ген, кодирующий часть рибосомы — ген 16S рРНК, а у эукариот — фрагменты геномов митохондрий или пластид: они накапливают больше замен, у них менее запутанное наследование, чем у ядерной ДНК, а ещё они присутствуют в клетке во множестве копий — значит, выделять и секвенировать их проще. Это особенно важно, если жизнь потрепала образец, и часть ДНК успела деградировать.
Известные последовательности баркодов можно найти, например, в библиотеке проекта BOLD (Barcode of Life Data System). Это самая крупная из подобных баз данных: к апрелю 2024 года она содержала баркоды для 365 тысяч видов живых организмов. Есть и более специализированные проекты: например, библиотека GreenGenes содержит баркоды для бактерий, а Diat.barcode — для коллекции диатомовых водорослей (это ещё один любимый класс растений у криминалистов — они тоже микроскопические, космически разнообразные по форме и покрыты панцирем из диоксида кремния). Баркодирование, конечно, применяют не только криминалисты: с его помощью идентифицируют патогены, находят новые виды, следят за биоразнообразием и состоянием экосистем, проверяют составы продуктов и растительных препаратов, так что библиотеки баркодов собираются совместными усилиями разных специалистов.
Можно ли верить «показаниям» пыльцы и грибов
Чем больше разных видов организмов обнаруживается в образце, тем сильнее в среднем сужается регион поиска. К счастью, технологии NGS (next generation sequencing) позволяют считывать баркоды буквально из грязи, получая информацию о видовой принадлежности всей водорослевой, например, или бактериальной ДНК. Такую совокупную ДНК называют метагеномом, а подход — метабаркодированием. В криминалистике он пока применяется не очень активно, но предварительные исследования вдохновляют: с помощью алгоритма машинного обучения Random Forest по бактериальному метагеному свежих следов можно определить, где гулял любитель тащить грязь в дом [8], а по составу кишечной микробиоты жителей Китая — прикинуть, на какой широте они живут [9].
Недавно китайские криминалисты применили метабаркодирование, чтобы опознать тело женщины [10]. В местных списках пропавших её не было; поиск по ДНК, по отпечаткам пальцев и другим приметам тоже ничего не дал. Тогда криминалисты заглянули к ней в лёгкие. Дело в том, что небольшая часть пыльцевых зёрен, попавших в дыхательные пути, остаётся в них навсегда, так что человек, долгое время проживший в одном районе, оказывается «помечен» пыльцой окрестных растений. Сопоставив образцы, полученные из лёгких, с географическим распределением флоры Китая, криминалисты смогли сузить поиски до двух провинций — и в итоге женщину удалось найти в списках пропавших в одной из них.
Насколько же точен этот метод? Чтобы проверить, авторы одного из исследований собрали в четырёх разных точках США 42 пробы пыли и попытались с помощью метабаркодирования восстановить регион её происхождения. Для этого карту США разбили на ячейки площадью 250 квадратных километров, и для каждой ячейки подсчитали, какой процент таксонов из тех, что обнаружены в пробе пыли, встречается на этой территории. Для каждой пробы получилась вот такая карта (Рисунок 2).
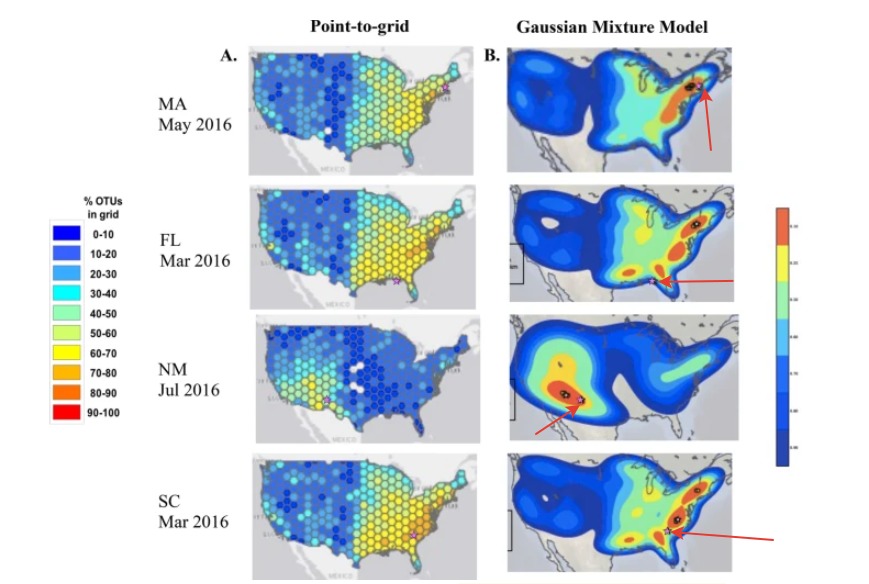
Рисунок 2. Результаты геолокации по пыльце. Стрелкой указана настоящая локация образца [11]
В «правильной» ячейке процент таксонов почти всегда был максимальным, но похожих ячеек оказалось много, так что точность геолокации получилась невысокой — около 600 км [11]. Информацию о том, где что растёт, авторы брали из базы данных проекта BISON, который каталогизирует флору и фауну Соединённых Штатов. Точность результата зависела как от времени сбора образцов, так и от региона: зимние образцы содержали пыльцу меньшего числа видов и потому хуже поддавались геолокации; разрешение метода также было ниже там, где большие пространства заняты относительно гомогенной флорой.
Исследователям, которые проанализировали с помощью нейронных сетей метагеномы грибов, бактерий и архей, геолокация удалась несколько лучше, чем тем, кто ориентировался на пыльцу [12]. Они взяли образцы пыли, собранной волонтёрами в рамках проекта Wild life of our homes (волонтёры собирали пыль с верхнего наличника внешней двери своего дома — а может, и чужого). На сей раз для половины из 1300 пыльных образцов, собранных в США, предсказанное расположение отличалось от настоящего менее чем на 100 километров. Когда тот же метод опробовали на пыли, собранной в разных странах, то оказалось, что для определения страны с точностью 90% достаточно одних грибов (точнее, метабаркодирования грибов).
Конечно, для геолокации по микробиомам необходимы постоянно обновляющиеся микробиомные карты, аналогичные картам распространения флоры, таким как BISON или iNaturalist. Зачатки таких карт уже существуют: например, проект MetaSUB c 2013 года собирает городские микробиомы по всему земному шару [13]. И хотя метод геолокации с помощью метабаркодирования ещё не получил широкого распространения, надеемся, что однажды карманные секвенаторы станут доступнее смартфонов, — и тогда вы сможете отследить по микробиому весь путь, который прошли кофейные зёрна, прежде чем добраться до вашей кофемолки.
Источники
- Mildenhall D. Civil and criminal investigations. The use of spores and pollen [Электронный ресурс ] // SIAK-Journal − Zeitschrift für Polizeiwissenschaft und polizeiliche Praxis. 2008. № 4. Pp. 35-52. DOI: 10.7396/2008_4_E. URL: http://dx.doi.org/10.7396/2008_4_E (дата обращения 11.04.2024).
- Suchan T., Talavera G., Sáez L., Ronikier M., Vila R. Pollen metabarcoding as a tool for tracking long-distance insect migrations [Электронный ресурс ] // Mol Ecol Resour. 2019. № 19. Pp. 149–162. DOI: 10.1111/1755-0998.12948. URL: https://onlinelibrary.wiley.com/doi/10.1111/1755-0998.12948 (дата обращения 11.04.2024).
- Goodman F. J. et al. PIGLT: A Pollen Identification and Geolocation system for forensic applications [Электронный ресурс ] // 2015 IEEE International Symposium on Technologies for Homeland Security (HST). Waltham, MA, USA, 2015. Pp. 1-7. DOI: 10.1109/THS.2015.7225271. URL: https://ieeexplore.ieee.org/document/7225271 (дата обращения 11.04.2024).
- Hesse M., Halbritter H., Heigl H., Auer W. 2021. Salix caprea. In: PalDat — A palynological database. https://www.paldat.org/pub/Salix_caprea/306378; accessed 2024-04-22.
- Halbritter H., Sam S., Weber M., Auer W. 2020. Alnus glutinosa. In: PalDat — A palynological database. https://www.paldat.org/pub/Alnus_glutinosa/303762; accessed 2024-04-22.
- Halbritter H. 2016. Picea abies. In: PalDat — A palynological database. https://www.paldat.org/pub/Picea_abies/302357; accessed 2024-04-22.
- Halbritter H., Sam S., Heigl H. 2020. Syringa vulgaris. In: PalDat — A palynological database. https://www.paldat.org/pub/Syringa_vulgaris/304333; accessed 2024-04-22.
- Zhang J., Yu D., Wang Y., Shi L., Wang T., Simayijiang H., Yan J. Tracing recent outdoor geolocation by analyzing microbiota from shoe soles and shoeprints even after indoor walking [Электронный ресурс ] // Forensic Science International: Genetics. 2023. Volume 65. DOI: 10.1016/j.fsigen.2023.102869. URL: https://www.sciencedirect.com/science/article/pii/S1872497323000443 (дата обращения 11.04.2024).
- He Y., Wu W., Zheng H. M. et al. Regional variation limits applications of healthy gut microbiome reference ranges and disease models [Электронный ресурс ] // Nat Med. 2018. № 24. Pp. 1532–1535. DOI: 10.1038/s41591-018-0164-x. URL: https://www.nature.com/articles/s41591-018-0164-x (дата обращения 11.04.2024).
- Chen Q., Liu M., Xu C. et al. Potential of plant DNA information in determining the provenance and identify of unknown victims [Электронный ресурс ] // Forensic Science International. 2023. Volume 350. DOI: https://doi.org/10.1016/j.forsciint.2023.111786. URL: https://www.sciencedirect.com/science/article/abs/pii/S0379073823002360?via%3Dihub (дата обращения 11.04.2024).
- Lennartz C., Kurucar J., Coppola S. et al. Geographic source estimation using airborne plant environmental DNA in dust [Электронный ресурс ] // Scientific Reports. 2021. № 11. DOI: 10.1038/s41598-021-95702-3. URL: https://www.nature.com/articles/s41598-021-95702-3 (дата обращения 11.04.2024).
- Neal S. Grantham, Brian J. et al. Global Forensic Geolocation with Deep Neural Networks [Электронный ресурс ] // Journal of the Royal Statistical Society Series C: Applied Statistics, August 2020, Volume 69, Issue 4, Pp. 909–929. DOI: doi.org/10.1111/rssc.12427.
- Danko D, et al. A global metagenomic map of urban microbiomes and antimicrobial resistance. [Электронный ресурс ] // Cell. 2021;184:3376–3393.e17. DOI: 10.1016/j.cell.2021.05.002.


