Плагиат — двигатель эволюции
Все живые существа делают это — заимствуют чужие тексты. Кто-то копирует часть курсовой, а кто-то идёт дальше и заимствует чужую ДНК. Геном, то есть закодированный в ДНК план организма, обычно наследуется «по вертикали», от предков. Но вдобавок можно присвоить гены неродственных — и иногда весьма непохожих — организмов. Такой генетический плагиат называется горизонтальным переносом генов (horizontal gene transfer, HGT). Можно сказать, что HGT — это естественный путь создания ГМО.
Например, бактерии при стрессе могут целенаправленно захватывать чужую ДНК в надежде, что там окажется закодировано что-нибудь спасительное: насос, который удалит из клетки антибиотик, или фермент, который позволит переваривать новый субстрат, когда вся привычная еда закончилась. Это быстрее и проще, чем изобретать оригинальное решение. Так распространяются полезные умения и даже возникают новые адаптации.
У бактерий обмен генами и целыми их комплектами настолько распространён, что схема родственных связей между разными видами — филогенетическое дерево — при добавлении информации о переносах превращается из дерева в сеть (Рис. 1).
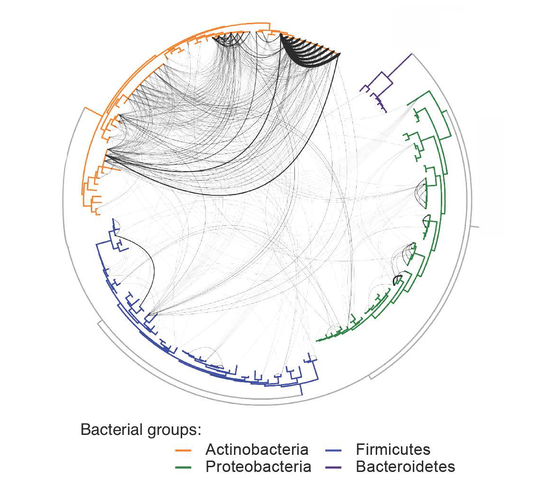
Рис. 1. Горизонтальный перенос генов между обитателями сырной корки. На окружности показано филогенетическое дерево; цветами отмечены разные отделы бактерий. События горизонтального переноса показаны чёрными дугами (чем больше генов одна бактерия позаимствовала у другой, тем толще дуга) [1]
Но и в остальных царствах живого горизонтальный перенос тоже случается. Недавно обнаружилось, что для насекомых это вполне обычное дело: учёные нашли признаки заимствований в 88% из 192 исследованных геномов, причём в среднем на геном приходилось по семь чужеродных вставок. Большая их часть была получена от бактерий, но встречались также грибные, растительные и вирусные гены [2]. А у бделлоидных коловраток (мелких многоклеточных, обитающих в пресной воде или мхе), самых усердных плагиаторов среди животных, заимствования составляют целых 10% генома [3].
Ген может кардинально изменить судьбу своего нового владельца — например, позволить ему захватить сушу. Растения когда-то переняли у почвенных бактерий и грибов гены, которые помогают синтезировать лигнин. Этот полимер накапливается при одревеснении и придает растениям прочность, а главное — формирует проводящую систему, «водопровод», без которого на суше не вырасти выше мха [4]. Приматы же благодаря HGT обзавелись своеобразной, крайне инвазивной плацентой (а вместе с ней, по-видимому, и менструациями [5]): оболочечные белки древнего вируса, который заражал предков человека, теперь помогают эмбриону внедряться в ткани матки и защищают его от материнского иммунитета [6].
Зачем искать плагиат в геноме
Выявлять генетические заимствования полезно: это помогает предсказывать (насколько возможно) дальнейшую эволюцию. Теперь известно, что патогенные бактерии, склонные к горизонтальному переносу генов, могут приобрести устойчивость к антибиотику, если найдут, у кого скопировать нужный ген. Определив проблему, можно с ней бороться: например, пробовать «лечить» микробиоту от генов устойчивости к антибиотикам [7].
Есть и другая причина изучать горизонтальный перенос: он запутывает схему родственных связей между организмами — филогению. А филогению важно знать для решения самых разных задач: например для ответа на вопрос, какие варианты гена приводят к заболеванию. Восстанавливают её с помощью сравнения геномов: в первом приближении, чем больше похожи последовательности геномов, тем более близкими родственниками они приходятся друг другу. Но заимствованные фрагменты генома имеют совсем другую эволюционную историю, а значит, искажают картину наследования. Так что биоинформатикам приходится сначала искать плагиат и исключать его из анализа, а потом уже строить филогению.
Как найти в геноме заимствования
Итак, хотим ли мы изучать происхождение древних генов или современную эволюцию патогенов — нам нужно уметь находить в геноме «плагиат». У учёных есть для этого два типа методов: филогенетические и параметрические. Филогенетические методы сравнивают эволюционные истории разных генов — прямо или косвенно, а параметрические анализируют сами тексты: они ищут гены, которые отличаются от остального генома частотами нуклеотидов («букв», из которых состоят ДНК и РНК) или их сочетаний.
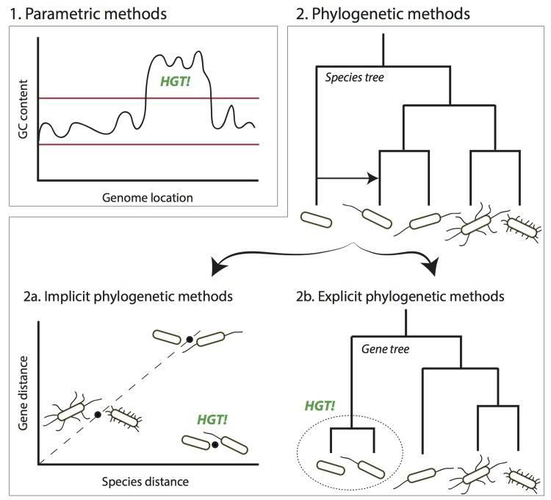
Рис. 2.1. Параметрические методы сравнивают последовательности генов. Ген, позаимствованный у другого организма, сохраняет характерные для этого организма частоты нуклеотидов. Если в геноме нашелся участок с нетипичными частотами — возможно, здесь случился горизонтальный перенос. Филогенетические методы прямо (2.2b) или косвенно (2.2a) сравнивают эволюционные истории генов, то есть филогенетические деревья, построенные на основании их последовательностей. Стрелка на рисунке 2.2 указывает направление горизонтального переноса. 2.2a. Горизонтальный перенос можно вычислить по неожиданно малому расстоянию между генами далеких родственников. 2.2b. А ещё филогенетическое дерево, построенное для одного из генов, отличается от дерева видов. Сравнивая топологии двух деревьев, можно понять, что именно однохвостая бактерия позаимствовала ген бесхвостой бактерии, а не наоборот [8]
Простейшие филогенетические методы поиска HGT немного похожи на поиск плагиата в тексте. Предположим, мы заподозрили, что некоторый ген попал в геном бактерии в результате горизонтального переноса. Мы можем обратиться к базе данных Genbank, в которой хранятся геномы более 500 000 разных организмов, поискать в ней последовательности, похожие на интересующий нас ген, и посмотреть, кому они принадлежат. Для этого обычно используется инструмент BLAST: он находит в базе все похожие фрагменты и для каждой находки указывает, насколько совпадение близкое и насколько оно статистически значимо. Близость совпадения определяется по метрике, напоминающей редакционное расстояние Левенштейна в автоматической обработке языка (NLP) — минимальное число однобуквенных вставок, удалений или замен, превращающих одно слово в другое. Значимость определяется по e-value — ожидаемому числу столь же близких или даже лучших совпадений, которые отыщутся среди того же числа случайных последовательностей.
Но в отличие от поиска плагиата, здесь статистически значимая находка сама по себе ещё не указывает на горизонтальный перенос: ведь функционально похожие гены разных организмов — в отличие от текстов разных авторов — чаще всего объединены общим «вертикальным» происхождением, поэтому их последовательности будут похожи по совершенно «законным» причинам. Под подозрение попадают только те гены, которые больше похожи на гены далёких родственников нашей бактерии, чем на гены её близких родственников (Рис. 2.2а) — а то и вовсе у близких не встречаются. Можно, например, определить для каждого гена «индекс чужеродности» [3], сравнив e-value самого похожего гена, найденного у близких родственников (скажем, внутри того же класса бактерий), с e-value самых похожих «неродственных» генов (принадлежащих любым другим организмам за пределами класса).
Примерно так исследователи обнаружили [9], что бактерии, обитающие в кишечнике у японцев, позаимствовали у морских бактерий гены ферментов для переваривания водорослей нори. Изначально учёные и не собирались исследовать HGT, они просто искали ферменты, которые способны разрушать клеточную стенку водорослей.
Разумеется, они искали их у морских бактерий, которые на этих водорослях живут и ими питаются. А когда нашли, то решили посмотреть, у кого ещё есть похожие гены. BLAST дал неожиданный ответ: они есть у бактерии Bacteroides plebeius, которая живёт вовсе даже не в море, а в кишечнике человека!
Пришлось учёным всё-таки заняться изучением HGT. Во-первых, они убедились, что у близких родственников B. plebeius, да и вообще ни у каких других наземных бактерий, похожих генов нет. Во-вторых, они проверили кишечных бактерий жителей США и выяснили, что американские B. plebeius, в отличие от японских, не помогают своим хозяевам переваривать водоросли. Это подтвердило гипотезу, что кишечные бактерии получили водорослеядный ген именно с помощью горизонтального переноса — от морских бактерий, в изобилии съедаемых японцами вместе с сырыми нори.

Рис. 3. Поедая сырые водоросли нори, японцы получили от морских бактерий способность переваривать водоросли нори. К сожалению, с обработанными нори из магазина, такими, как на картинке, этот фокус не пройдёт. Источник: Википедия
Здесь плагиат бросался в глаза. Но что если перенос генов произошёл между близкими родственниками? Или ген в ходе эволюции неоднократно терялся и приобретался, и нашлось несколько возможных доноров? В сложных случаях может помочь детальный филогенетический анализ (Рис. 2.2b): нужно построить филогенетическое дерево, основываясь на последовательности подозрительного гена, и сравнить его с истинной эволюционной историей видов. Если две истории заметно расходятся, это может указывать на горизонтальный перенос.
Но как же узнать истинную историю? Есть так называемые гены домашнего хозяйства, критически важные для организма, которые меняются очень неохотно, например те, что отвечают за синтез ДНК и белков. Единственная мутация в таком гене может оказаться смертельной. Тем более невероятно, чтобы организм пережил «пересадку» гена от другого вида, поэтому мы верим, что гены домашнего хозяйства наследуются только по вертикали и рассказывают настоящую эволюционную историю вида.
Здесь читатель может заметить, что ему сначала предложили искать заимствования во имя построения правильной филогении, а затем посоветовали построить филогению, чтобы найти заимствования. Никакого обмана: филогения основывается на изменчивых участках, которых в генах домашнего хозяйства мало, а значит, разрешение у нашего канонического дерева получится низким. Если найти и исключить плагиат, на оставшихся генах можно построить более детальную филогению.
Стилометрия по-биологически
Второй подход к поиску горизонтального переноса, параметрический, похож на стилометрию. ДНК — полимер, состоящий из четырёх возможных нуклеотидов, обозначаемых буквами A, T, G и С. Часть ДНК составляют гены, в которых закодированы белки: последовательность из трёх нуклеотидов (триплет) кодирует одну из 21 аминокислоты. Таким образом, геном — своего рода текст. Авторы отличаются друг от друга лексикой или синтаксисом, геномы же имеют разный нуклеотидный состав, и заимствованный ген будет хранить следы нуклеотидных предпочтений своего прежнего владельца.
В одних случаях могут различаться частоты самих нуклеотидов (Рис. 2.1). Например, у бактерии Candidatus Zinderia insecticola частота нуклеотидов G и C в геноме составляет 13,5 %, а у Anaeromyxobacter dehalogenans, находящейся на другом краю этого спектра, — 75 % [10]. В других случаях, исходных владельцев гена, как и авторов текста, можно идентифицировать по тому, какие синонимы они раз за разом выбирают из ряда возможных. Да, в генетическом коде тоже есть синонимы: большинство аминокислот кодируется несколькими разными триплетами. Например, аминокислота глицин может быть записана в ДНК четырьмя способами: как GGA, GGT, GGC или GGG. Многие живые существа предпочитают одни триплеты другим, причём предпочтения у каждого свои.
Наконец, в качестве характерных признаков можно использовать не только кодирующие триплеты, но и вообще любые короткие k-меры, то есть последовательности нуклеотидов длины k. Характерные для генома частоты нуклеотидов, триплетов или k-меров — их можно вычислить, опять-таки, по генам домашнего хозяйства — они называются геномной подписью.
У параметрического подхода есть несколько проблем. Во-первых, он не будет работать, если у реципиента и донора похожие подписи. Во-вторых, геномная подпись донора со временем стирается: на новом месте мутации происходят и закрепляются уже в соответствии с предпочтениями другого хозяина, поэтому метод подходит только для поиска недавних событий. Кроме того, геномы неоднородны по нуклеотидному составу, даже если никакого плагиата в них нет. Например, часто используемые синонимы нравятся организму тем, что они быстрее прочитываются рибосомой при синтезе белка. Если белка нужно много, этот выигрыш в скорости важен, и естественный отбор фиксирует в гене «быстрые» синонимы. Но если скорость синтеза белка значения не имеет, то соответствующий ген менее разборчив в синонимах — и наивный параметрический метод мог бы ошибочно принять его за плагиат.
Параметрический подход был незаменим, пока известных геномов было совсем мало. Новые технологии сильно удешевили секвенирование и чуть не отправили метод на свалку истории. Но они же подбрасывают новые задачи на «определение авторства», для которых параметрический метод отлично подходит: например, отделить друг от друга фрагменты геномов, принадлежащих разным неизвестным организмам. Дело в том, что самые популярные приборы для секвенирования вместо готового текста выдают множество пересекающихся фрагментов длины 100–600 нуклеотидов (чтений), из которых геном ещё нужно собрать, как пазл. Если в секвенируемом образце есть ДНК нескольких видов микроорганизмов — например, это образец почвы — то собрать неизвестные геномы становится ещё сложнее: ведь непонятно, какие кусочки к какому геному относятся. Если сборку одного генома часто сравнивают с попыткой восстановить содержание газеты из разорванной на клочки стопки одинаковых номеров, то в случае метагеномных исследований (когда секвенируются все геномы образца разом) на клочки порвано уже содержимое целого газетного ларька (исключая продавщицу). Разница в геномных подписях помогает разделить перемешанные пазлы, то есть сгруппировать фрагменты, относящиеся к тому или иному геному, — и после этого уже можно собирать отдельные геномы [11].
Тёмная сторона горизонтального переноса генов
Генам, безусловно, выгодно встраиваться в новые последовательности. А вот выгоден ли перенос самим организмам-плагиаторам? Некоторые бактерии начинают охотиться на чужую ДНК, едва уловив сигналы бедствия от соседей и не дожидаясь, пока им самим станет плохо [12]. Они, очевидно, тоже в выигрыше.
Но в целом копирование чужих генов — рискованное мероприятие. Во-первых, что одной бактерии хорошо, то другой — смерть: полезность гена зависит от того, в какое окружение он попадает. Во-вторых, место для встраивания выбирается почти случайно, поэтому вставка может задеть другой ген и сломать его или нарушить его регуляцию. На случайности процесса даже основан ещё один метод поиска горизонтального переноса. У бактерий гены часто формируют функциональные кластеры: гены белков, выполняющих общую задачу, расположены в геноме друг за другом. А переехавший ген выбивается из контекста, как копипаста в плохом реферате.
Наконец, к горизонтальному переносу особенно склонны так называемые мобильные элементы, «слова-паразиты», которые способны размножаться внутри генома и перепрыгивать с места на место, засоряя его. У человека из таких элементов состоит примерно половина размера генома. Большая часть мобильных элементов, к счастью, давно потеряла подвижность, но выжившие могут вызывать заболевания своими неудачными прыжками [13, 14].
По мере того как совершенствуются методы поиска плагиата, усложняются научные вопросы. Можно ли предотвратить или стимулировать горизонтальный перенос? И как меняется эволюция изменённых таким образом организмов? Всё это предстоит прояснить биологам в будущем.
Использованные источники
- Bonham K. S., Wolfe B. E., Dutton R. J. Extensive horizontal gene transfer in cheese-associated bacteria // eLife Sciences Publications, Ltd : сайт. URL: https://elifesciences.org/articles/22144 (дата обращения: 14.08.2024).
- Ли Яю, Лю Ч., Лю Ч. и др. HGT is widespread in insects and contributes to male courtship in lepidopterans // Национальная медицинская библиотека Национального центра биотехнологической информации США : офиц. сайт. URL: https://www.ncbi.nlm.nih.gov/pmc/articles/PMC9357157 (дата обращения: 14.08.2024).
- Gladyshev E. A. et al. Massive Horizontal Gene Transfer in Bdelloid Rotifers // Science, 2008. № 320. Pp/ 1210-1213(2008). DOI:10.1126/science.1156407 URL: https://www.science.org/doi/10.1126/science.1156407 (дата обращения: 14.08.2024).
- Emiliani G., Fondi M., Fani R. et al. A horizontal gene transfer at the origin of phenylpropanoid metabolism: a key adaptation of plants to land // Biol Direct. 2009. Vol. 4, issue 7 (2009). DOI: 10.1186/1745-6150-4-7. URL: https://biologydirect.biomedcentral.com/articles/10.1186/1745-6150-4-7 (дата обращения: 14.08.2024).
- Thomas V. G. The Link Between Human Menstruation and Placental Delivery: A Novel Evolutionary Interpretation: Menstruation and fetal placental detachment share common evolved physiological processes dependent on progesterone withdrawal // Bioessays. 2019. № 41(6). DOI: 10.1002/bies.201800232. URL: https://pubmed.ncbi.nlm.nih.gov/31119755/ (дата обращения: 14.08.2024).
- Шкроб М. «Когда б вы знали, из какого сора…» [Электронный ресурс] // Химия и жизнь. 2009. №10. URL: https://elementy.ru/nauchno-populyarnaya_biblioteka/431045/Kogda_b_vy_znali_iz_kakogo_sora (дата обращения: 14.08.2024).
- Buckner M. M. C, Ciusa M. L., Piddock L. J. V. Strategies to combat antimicrobial resistance: anti-plasmid and plasmid curing // FEMS Microbiol Rev. 2018. Vol. 42(6). Pp. 781-804. DOI: 10.1093/femsre/fuy031. URL: https://www.ncbi.nlm.nih.gov/pmc/articles/PMC6199537 / (дата обращения: 14.08.2024).
- Ravenhall M., Škunca N., Lassalle F., Dessimoz C. Inferring horizontal gene transfer // PLoS Comput Biol. 2015. Vol.11(5). DOI: 10.1371/journal.pcbi.1004095. URL: https://www.ncbi.nlm.nih.gov/pmc/articles/PMC4462595/ (дата обращения: 14.08.2024).
- Hehemann J.-H., Correc G, Barbeyron T, Helbert W, Czjzek M, Michel G. Transfer of carbohydrate-active enzymes from marine bacteria to Japanese gut microbiota // Nature. 2010 Apr 8;464(7290):908-12. doi: 10.1038/nature08937. PMID: 20376150.
- Almpanis A. et al. Correlation between bacterial G+ C content, genome size and the G+ C content of associated plasmids and bacteriophages // Microbial genomics. 2018. Vol. 4. Issue 4. URL: https://www.microbiologyresearch.org/content/journal/mgen/10.1099/mgen.0.000168 (дата обращения: 14.08.2024).
- Wu Y. W., Simmons B. A., Singer S. W. MaxBin 2.0: an automated binning algorithm to recover genomes from multiple metagenomic datasets // Bioinformatics. 2016. Vol. 32. Issue 4. Pp. 605–607.URL: https://academic.oup.com/bioinformatics/article/32/4/605/1744462 (дата обращения: 14.08.2024).
- Prudhomme M. et al. Pneumococcal competence is a populational health sensor driving multilevel heterogeneity in response to antibiotics // Nature Communications. 2024. Vol. 15. Issue 1. URL: https://www.nature.com/articles/s41467-024-49853-2 (дата обращения: 14.08.2024).
- Kemp J. R., Longworth M. S. Crossing the LINE toward genomic instability: LINE-1 retrotransposition in cancer // Frontiers in chemistry. 2015. Vol. 3. URL: https://www.frontiersin.org/journals/chemistry/articles/10.3389/fchem.2015.00068/full (дата обращения: 14.08.2024).
- Мобильные генетические элементы: подборка статей [Электронный ресурс] // Научный проект «Биомолекула»: сайт. URL: https://biomolecula.ru/themes/mge (дата обращения: 14.08.2024).