Долгое время археологи и антропологи исследовали древние общества, опираясь на собственные методы. Для датировки находок анализировались надписи, типы объектов, культурные слои, химический состав, остаточная намагниченность, содержание радиоактивных изотопов углерода. Однако новые методы молекулярной биологии существенно дополнили подход к изучению древних цивилизаций. Благодаря палеогенетикам и биоинформатикам можно проводить генетический анализ древней ДНК и воссоздавать по нему истории наших предков, а порой даже опровергать теории, существующие на протяжении многих лет.
О том, как междисциплинарный подход в науке помогает узнать ранее неизвестные детали о древнем геноме и открывает новые перспективы для изучения самого далекого прошлого человека, рассказывает Андрей Макашов, преподаватель специалитета по биоинженерии и биоинформатике петербургского Политеха.
Как в археологию пришёл анализ ДНК
Междисциплинарность позволяет учёным объединять знания и методы исследований для решения научных задач. Поэтому исследователи всё чаще объединяются в группы, чтобы получать новые результаты на стыке разных дисциплин. Так произошло и с изучением древнего человека. В течение многих десятилетий археологи полагались на археологические данные, формулируя гипотезы на основе своих классических методов датировки.
Всё изменилось, когда в археологию ворвалась палеогенетика — новая область науки, которая изучает древнюю ДНК (палеоДНК), содержащуюся в биологических останках организмов, и анализирует её на основе археологических, палеонтологических и музейных образцов [1]. Основоположник палеогенетики — Сванте Паабо, который в 1985 году представил первые результаты анализа древней ДНК египетской мумии ребёнка возрастом 2400 лет [2].

Сванте Паабо. Источник
При этом первое исследование по анализу ДНК из останков умерших существ, получившее мировой резонанс, было опубликовано годом ранее другой командой учёных под руководством Аллана Уилсона. Его группа извлекла ДНК из музейного образца квагги — зеброподобного вида, вымершего в 1883 году, и представила результаты в журнале Nature [3].
Последние 15–20 лет палеогенетика быстро прогрессирует благодаря росту молекулярно-генетических технологий и особенно внедрению методов современного секвенирования ДНК. Они позволили получать больший объём информации из меньшего количества исходного образца [4].
Сегодня палеогенетика находится на стыке молекулярной биологии, археологии и биоинформатики и стремительно развивается. В 2022 году Сванте Паабо был удостоен Нобелевской премии по физиологии или медицине «за открытия в области геномов вымерших гоминидов и эволюции человека».
Археологи-палеогенетики-биоинформатики
Археологи проводят раскопки, находят древние артефакты и останки и передают часть из них палеогенетикам. ДНК лучше всего сохраняется при низкой влажности и минимальных температурах, и в России благодаря климату обнаруживают большое количество палеоматериала, пригодного для генетических исследований.
Палеогенетики выделяют генетический материал из зубов, костей, волосяных фолликул и других сохранившихся тканей. При этом уже есть случаи, когда учёным удавалось прочесть ДНК из осадочных пород [5] и из костяной подвески, принадлежащей древней женщине [6].
Выделение генетического материала — сложный процесс, поскольку результат сильно зависит от степени деградации и контаминации (загрязнения) древней ДНК. Полученные фрагменты палеогенетики секвенируют, то есть определяют первичную последовательность нуклеотидов.
На заключительном этапе в работу вступают биоинформатики. Они используют специализированные программы для обработки данных расшифрованной палеоДНК. Результаты секвенирования содержат информацию о современной ДНК, случайно попавшей в образец, и нераспознанные фрагменты — генетический «мусор». Именно его сначала и удаляют специалисты. На этом этапе исключаются те последовательности, которые в дальнейшем могут привести к ошибкам. После обработки данных происходит сравнение древних геномов между собой или с современными ДНК-последовательностями.
Одна из стадий биоинформатического анализа ДНК — определение гаплотипов, то есть совокупностей генетических вариантов (аллелей), свойственных индивиду. Иногда исследуют только мутации в Y-хромосоме и в митохондриальной ДНК, то есть Y-хромосомный и митохондриальный гаплотипы. Y-хромосома передается только по мужской линии, а митохондрии — только по женской, генетический материал отца и матери не перемешивается (не рекомбинирует), поэтому такие гаплотипы удобно использовать для изучения генеалогии и популяционной генетики. Например, по анализу митохондриальной ДНК установлено время существования последнего общего предка кроманьонца и неандертальца, которое оценивается в 500 тыс. лет. Благодаря биоинформатике и палеогенетике из древней ДНК можно получать информацию для определения эволюционных связей между организмами и происхождения инфекционных болезней, изучения миграции населения и генетических адаптаций к культурным изменениям, описания черт внешности древних людей и их предрасположенности к заболеваниям. Хотя исследование современных популяционных частот тоже позволяет отследить некоторые доисторические события по косвенным признакам, древняя ДНК — это «окно» в прошлое, через которое можно подсмотреть правильный ответ.
Как палеогенетика доказала наше родство с неандертальцами
Один из главных результатов палеогенетических исследований — история родства между неандертальцами и современным человеком. Долгое время неандертальцы считались вымершим параллельным видом Homo. В 2010 году в журнале Science был опубликован анализ ДНК Homo neanderthalensis, извлеченной из трёх образцов костей, которые были найдены в пещере Виндия в Хорватии [7]. Это исследование впервые подтвердило, что неандертальцы и современные люди имеют общие гены, а любой европеоид наследует 1–2% генетического материала от неандертальцев [8].

Женщина-неандерталец, реконструкция внешности по черепу. Источник
Не менее интересными и значимыми стали палеогенетические результаты по найденному ранее неизвестному науке виду древнего человека — денисовцу, который проживал на территории современной Сибири. Учёные обнаружили фалангу пальца в отложениях Денисовой пещеры на Алтае. Денисовцы жили там примерно от 100 до 50 тыс. лет назад, а возможно, в какой-то период времени даже делили пещеру с неандертальцами. Сравнение найденного генома с другими известными показало, что обнаруженные останки принадлежали человеку, значительно отличающемуся по типу митохондриальной ДНК от неандертальца и современного человека [9]. Так, впервые в истории науки удалось определить вид древнего человека на основе анализа ДНК, а не по внешним характеристикам костей, черепа и т. п.
Ряд исследований показали: не только неандертальцы, но и денисовцы внесли свой вклад в генофонд современного человека. Например, стало известно, что жители тихоокеанского бассейна, включая австралийских аборигенов и обитателей Папуа, унаследовали около 5% генетического материала от денисовского человека [10], а население материковой части Азии — лишь 0,2% таких генов [11]. 80% населения, проживающего в высокогорном Тибете, вероятно, оказалось генетически адаптированным к низкому содержанию кислорода в воздухе благодаря гену, также унаследованному от денисовца [12].
Палеогенетики полагают, что неандертальцы, денисовцы и, возможно, другие древние группы, которые населяли Европу сотни тысяч лет назад, неоднократно пересекались и скрещивались с людьми современного типа. Это позволило увеличить генетическую изменчивость вида и лучше адаптироваться популяции к суровым условиям среды.
Древняя ДНК помогает отследить миграции
С помощью анализа палеоДНК изучают и перемещения населения в далёком прошлом. Палеогенетики могут определить, менялись ли древние общества на определённой территории в результате миграций или народ вёл оседлый образ жизни. Анализ древней митохондриальной ДНК европейских популяций эпохи неолита (новый каменный век) показал, что земледельцы из Ближнего Востока мигрировали в Европу, принеся с собой новые технологии выращивания культурных растений.
Чуть позже эту же теорию удалось подтвердить на основе секвенирования ядерного генома древних образцов [13]. По гипотезе авторов исследования генофонд Европы формировался из трёх древних популяций. Первая состояла из европейских охотников-собирателей, проживавших со времен палеолита. Вторая формировалась из ближневосточных земледельцев, которые переселились на территорию Европы и смешались с местными аборигенами. А третья — из охотников-собирателей, которые впервые были обнаружены из анализа древнего образца со стоянки Мальта в Сибири, за что и получили название «древние североевразийцы».
С ростом количества древних образцов, из которых можно выделить ДНК, увеличивался и объём исследований, по итогам которых стали говорить о двух массовых волнах миграции в Европу. Первая произошла в раннем неолите и связана с переселенцами-земледельцами с Ближнего Востока. Данные палеогенетиков по этой волне миграции пересекаются с выводами археологов и в целом признаются практически всеми специалистами. Вторая волна миграции в Европу возникла в эпоху позднего неолита и начала бронзового века. Её связывают с населением археологической ямной культуры, которое пришло из причерноморско-каспийских степей. У ямников основным видом хозяйства было скотоводство. Они мигрировали на протяжении нескольких поколений и значительно повлияли на генофонд европейцев. Но не все археологи и лингвисты соглашаются с выводами палеогенетиков по поводу этой волны миграции. Поэтому многие гипотезы до сих пор вызывают междисциплинарные споры.
Также миграции древних популяций можно восстанавливать по маршрутам распространения центров, где происходило одомашнивание сельскохозяйственных животных. Например, выявленная генетическая связь между современными и древними коровами Центральной Европы с ближневосточным происхождением дополнительно подтвердила теорию о том, что первые европейские землевладельцы в неолите были из региона Ближнего Востока [14].
Прояснить пути миграции древнего человека на территории современной России помогло изучение могильника, датированного 5197–4850 гг. до н. э., в окрестностях города Нальчик. Учёные извлекли древнюю ДНК из зуба, найденного на этом захоронении, и обнаружили, что примерно 5000 лет до н. э. на Северном Кавказе смешались генетические группы кавказских охотников-собирателей и представители раннего докерамического неолита, которые, вероятно, пришли из Северной Месопотамии — Загроса. Это население активно взаимодействовало с восточными охотниками-собирателями из степных регионов. Благодаря этой работе учёные подтвердили гипотезу о главных миграционных потоках из Ближнего Востока в Европу.
Происхождение и пути распространения болезней
Анализ палеоДНК также позволяет выявлять заболевания у древнего населения и генетическую предрасположенность к ним. Например, в Корее учёные исследовали мумию женщины XVII века и обнаружили сильную генетическую предрасположенность к атеросклеротическому сердечно-сосудистому заболеванию [15]. До активного развития палеогенетики информацию о болезнях выясняли по костным останкам антропологическими методами, но этого не всегда было достаточно, поскольку не все болезни оставляют следы на костях. Первым результатом палеогенетических исследований в этой области стала расшифрованная ДНК из зубов людей, найденных в массовом захоронении Марселя. Учёные в 1998 году обнаружили в образцах части генома бактерии Yersinia pestis — возбудителя чумы, тем самым подтвердив, что во Франции в конце XVI века была эпидемия [16]. Ещё один древний возбудитель чумы выделили из зубной пульпы людей, которые скончались в бронзовом веке и были погребены на территории современной Самарской области [17].
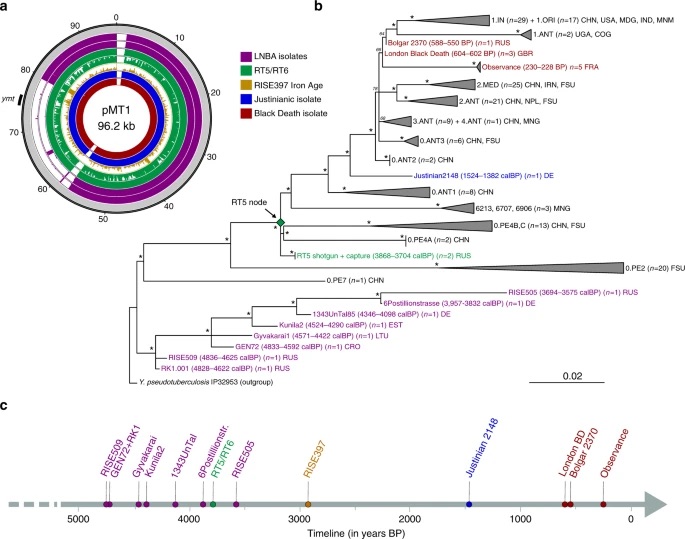
Филогенетическое дерево различных исторических штаммов чумы. Источник: [17]
В одном из самых последних исследований учёные проанализировали древнюю ДНК из костей и зубов 108 человек и у 17% обнаружили чуму. Все они жили на территории современной Дании и Швеции 5 тыс. лет назад. Это исследование, вероятно, указывает на то, что именно эпидемия чумы могла стать причиной сокращения населения в Европе в тот период [18]. Раньше предполагалось, что чума стала вызывать серьёзные эпидемии, когда научилась использовать блох в качестве переносчика. Анализ древней ДНК показал, что это не так: у большинства штаммов Y. pestis, относящихся к захоронениям 3–1 тысячелетий до нашей эры, нет мутаций, необходимых для успешного размножения внутри блох — хотя в бронзовом веке штаммы с такими мутациями уже существовали [17], распространились они позже.
Возбудители проказы менее изучены. Однако есть ряд работ: например, международный проект для исследования методов лечения проказы в Средние века в Европе. Учёные из Римского университета Сапиенца проанализировали зубной камень, который нашли на старых английских и французских кладбищах, и обнаружили следы имбиря. С большой вероятностью именно им и пытались лечить проказу. Эта болезнь — одна из самых древних, и во времена Средневековья была достаточно распространённой, что, возможно, связано с перемещениями крестоносцев.
Ещё один известный кейс — установление возбудителя болезни, которая выкосила 80% коренного ацтекского населения Центральной Америки после прихода европейских колонизаторов. Долгое время название болезни было известно только по хроникам самих ацтеков. Благодаря методам палеогенетики и секвенированию ДНК с кладбищ удалось установить, что ацтеков погубил брюшной тиф.
Палеогенетика — новый инструмент археолога
До интенсивного развития палеогенетики и биоинформатики множество гипотез, выдвинутых на основании археологических и антропологических данных, нельзя было точно проверить. С привлечением новой дисциплины это стало возможным. Иногда полученные палеогенетиками выводы совпадают с теориями археологов, историков и лингвистов, а порой, напротив, опровергают их.
По словам Вольфганга Хаака, руководителя группы отдела археогенетики Института эволюционной антропологии Макса Планка, «генетика вносит важный фрагмент в общий пазл. Не больше, но и не меньше». А руководитель лаборатории в ФИЦ «Фундаментальные основы биотехнологии» РАН Егор Прохорчук считает палеогенетику скорее прикладной наукой. «Мы — инструмент в руках археологов, — подчеркнул он. — Мы не хотим навязывать свои гипотезы. И я был бы очень аккуратен в использовании древней ДНК в исторических вопросах».
Палеогенетика, бесспорно, важна в исследованиях древних популяций, но она скорее играет роль звена, соединяющего разные научные направления в единый междисциплинарный подход.
Источники
- Eroshenko, G. & Batieva, E. & Kutyrev, Vladimir. (2023). Paleogenomics of the Plague Agent and Prospects for Paleogenomic Studies in Russia. Problems of Particularly Dangerous Infections. 13-28. 10.21055/0370-1069-2023-2-13-28.
- Pääbo, S. Molecular cloning of Ancient Egyptian mummy DNA. Nature 314, 644–645 (1985). https://doi.org/10.1038/314644a0.
- Higuchi, R., Bowman, B., Freiberger, M. et al. DNA sequences from the quagga, an extinct member of the horse family. Nature 312, 282–284 (1984). https://doi.org/10.1038/312282a0.
- Пилипенко А. С. Палеогенетика человека. Вавиловский журнал генетики и селекции, 2013, Т.17, № 4/2 c. 960.
- Viviane Slon et al.,Neandertal and Denisovan DNA from Pleistocene sediments.Science356,605-608(2017).DOI:10.1126/science.aam9695.
- Essel, E., Zavala, E. I., Schulz-Kornas, E., et al. Ancient human DNA recovered from a Palaeolithic pendant. Nature 618, 328–332 (2023). https://doi.org/10.1038/s41586-023-06035-2.
- 7. Richard E. Green et al., A Draft Sequence of the Neandertal Genome. Science 328, 710-722(2010).DOI:10.1126/science.1188021.
- Sankararaman S., Patterson N., Li H., Pääbo S., Reich D. (2012) The Date of Interbreeding between Neandertals and Modern Humans. PLOS Genetics 8(10): e1002947.https://doi.org/10.1371/journal.pgen.1002947.
- Reich D. et al. Genetic history of an archaic hominin group from Denisova Cave in Siberia //Nature. — 2010. — V. 468. — №. 7327. — P. 1053-1060.
- Reich, D. et al. The American Journal of Human Genetics, Volume 89, Issue 4, 516–528.
- Prüfer, K., Racimo, F., Patterson, N. et al. The complete genome sequence of a Neanderthal from the Altai Mountains. Nature 505, 43–49 (2014). https://doi.org/10.1038/nature12886.
- Huerta-Sánchez et al. (2014). Altitude adaptation in Tibetans caused by introgression of Denisovan-like DNA. Nature, 512(7513), 194–197.doi:10.1038/nature13408.
- Haak, W., Lazaridis, I., Patterson, N. et al. Massive migration from the steppe was a source for Indo-European languages in Europe. Nature 522, 207–211 (2015). https://doi.org/10.1038/nature14317.
- Edwards C. J., Bollongino R., Amelie Scheu A. et al. Mitochondrial DNA analysis shows a Near Eastern Neolithic origin for domestic cattle and no indication of domestication of European aurochs // Proc. Biol. Sci. 2007. V. 274. P. 1377–1385.
- Dong Hoon Shin. Paleogenetic study on the 17th century Korean mummy with atherosclerotic cardiovascular disease / Dong Hoon Shin [et al.] // PLOS ONE. — 2017. — No 12. — P. 1–14.
- Drancourt M., Aboudharam G., Signoli M., Dutour O., Raoult D. Detection of 400-year-old Yersinia pestis DNA in human dental pulp: an approach to the diagnosis of ancient septicemia. Proc Natl Acad Sci U S A. 1998 Oct 13;95(21):12637-40. doi: 10.1073/pnas.95.21.12637. PMID: 9770538; PMCID: PMC228830.
- Spyrou, M. A., Tukhbatova, R. I., Wang, CC. et al. Analysis of 3800-year-old Yersinia pestisgenomes suggests Bronze Age origin for bubonic plague. Nat Commun 9, 2234 (2018). https://doi.org/10.1038/s41467-018-04550-9.18. Seersholm, F. V., Sjögren, KG., Koelman, J. et al. Repeated plague infections across six generations of Neolithic Farmers. Nature (2024). https://doi.org/10.1038/s41586-024-07651-2.
- Seersholm, F. V., Sjögren, KG., Koelman, J. et al. Repeated plague infections across six generations of Neolithic Farmers. Nature (2024). https://doi.org/10.1038/s41586-024-07651-2.